1. 概述
1.1 聚类和分类
有人会觉得聚类就是分类,而其实在严格意义上,聚类与分类并不是一回事,两者有着很大的差异。
分类是按照已定的程序模式和标准进行判断划分,如男人和女人,在这里,你可以理解为这是分类/分组数据(注意了,不同的数据类型有不同的检验方法哟)。再比如,我们直接规定把数据的平均值作为中心轴,那么我们可以把这一组数据分为两个组别,即高于平均和等于平均的两个组,这也是分类。顺便扩展一下,我们后期会讲一期单基因GESA分析,用到的分组原理大概就是这个呢。因此,分类是按照固有或者某个标准将数据进行分组。
而聚类则分析,事先我们并不知道具体的划分标准,要靠算法进行判断数据之间的相似性,把相似的数据放在一起,也就是说聚类最关键的工作是:探索和挖掘数据中的潜在差异和联系。
在聚类的结论出来之前,我完全不知道每一类有什么特点,一定要根据聚类的结果通过人的经验来分析,看看聚成的这一类大概有什么特点。
1.2 聚类的方法
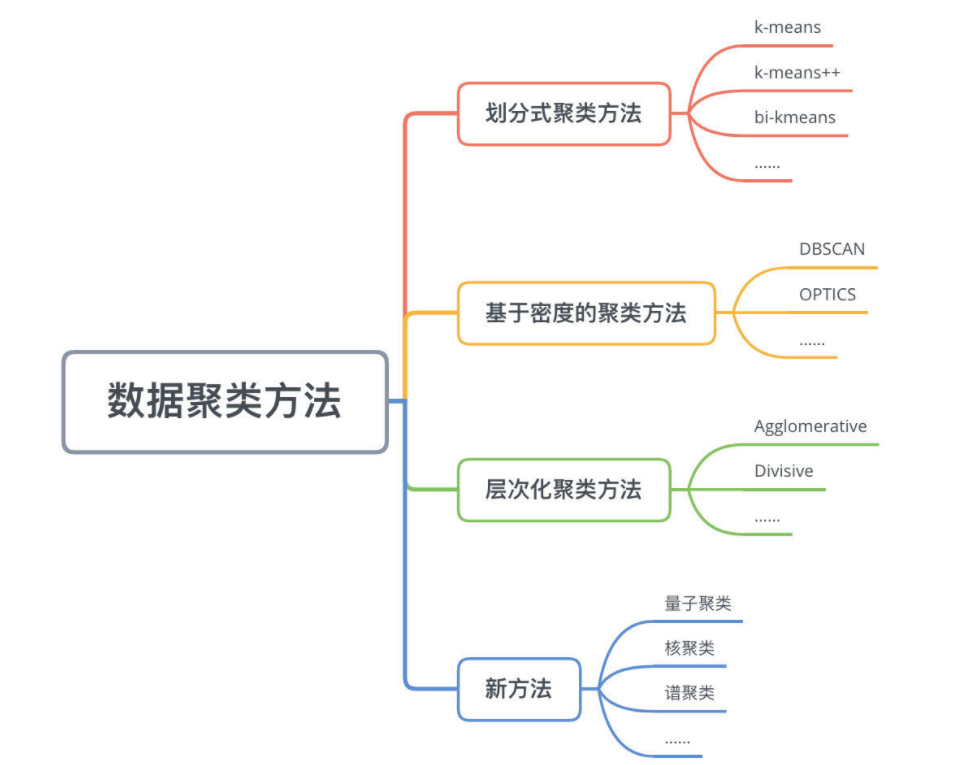
关于聚类的各种算法,推荐知乎的一篇文章点我学习,下例图片来源于知乎。
2 聚类在R语言中的实现
2.1 包的安装
这里,我们首先用到两个包,第一个是WGCNA,第二个是openxlsx,加载它们:
# Load the WGCNA package
library(openxlsx) # 导入Excel数据
library(WGCNA) # 画聚类图如果还没有安装这两个包,安装方法请移步我的另一篇博文用R语言如何画一张漂亮的热图。
2.2 数据读入
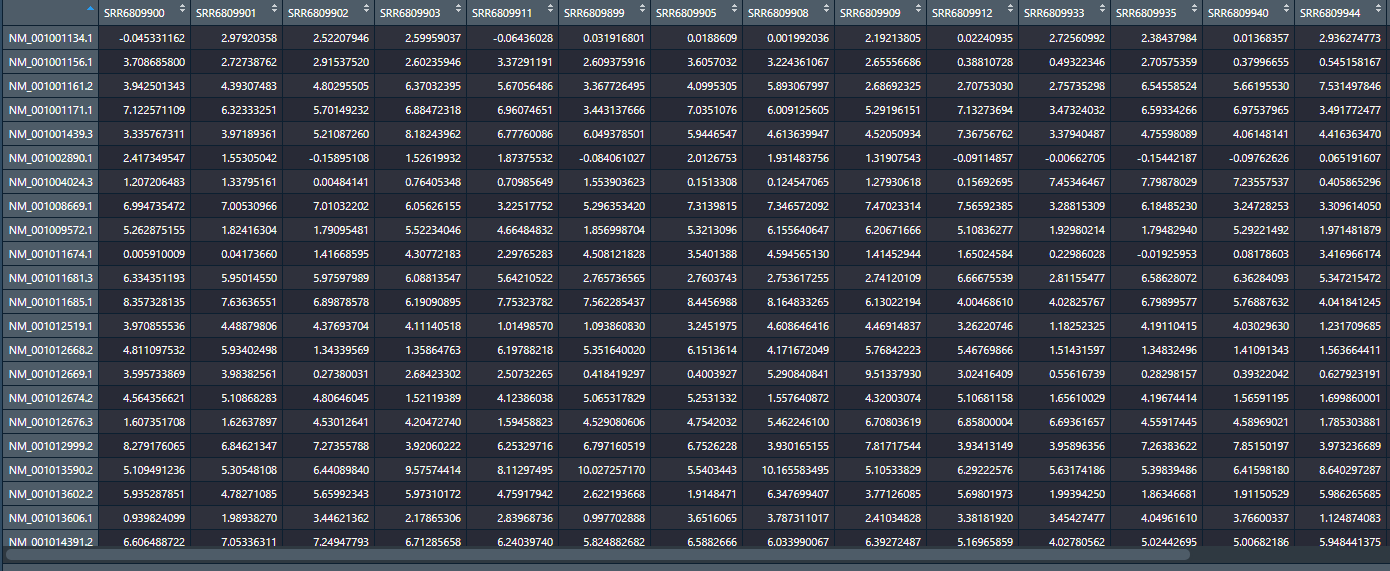
先上数据格式,见下图:
再上代码:
dat <- read.xlsx("filename.xlsx")将上述矩阵转置并转化为数据框
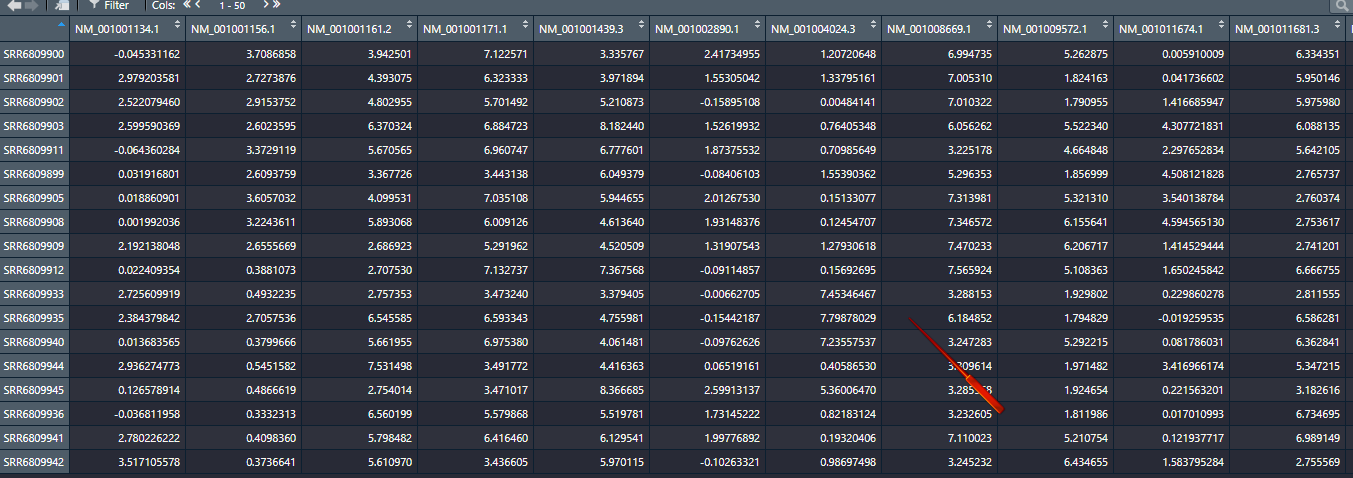
datExpr0 = as.data.frame(t(dat[,]))转置之后数据格式如下图,行名是样本名,列名是基因名:
画图
如下就可以画图啦
sampleTree = hclust(dist(datExpr0), method = "average") # 选择计算中心距方法
sizeGrWindow(16,9) # 设置画图框的大小
par(mar = c(4,4,4,4)) # 设置边距,根据实际情况选择
plot(sampleTree,
main = "Filemane",#图名字
sub="", # 标题名字
xlab="", # X轴名字
cex.lab = 1.5, # X轴Y轴名字大小
cex.axis = 1.5, #Y轴刻度大小大小
cex.main = 1.5) #标题大小
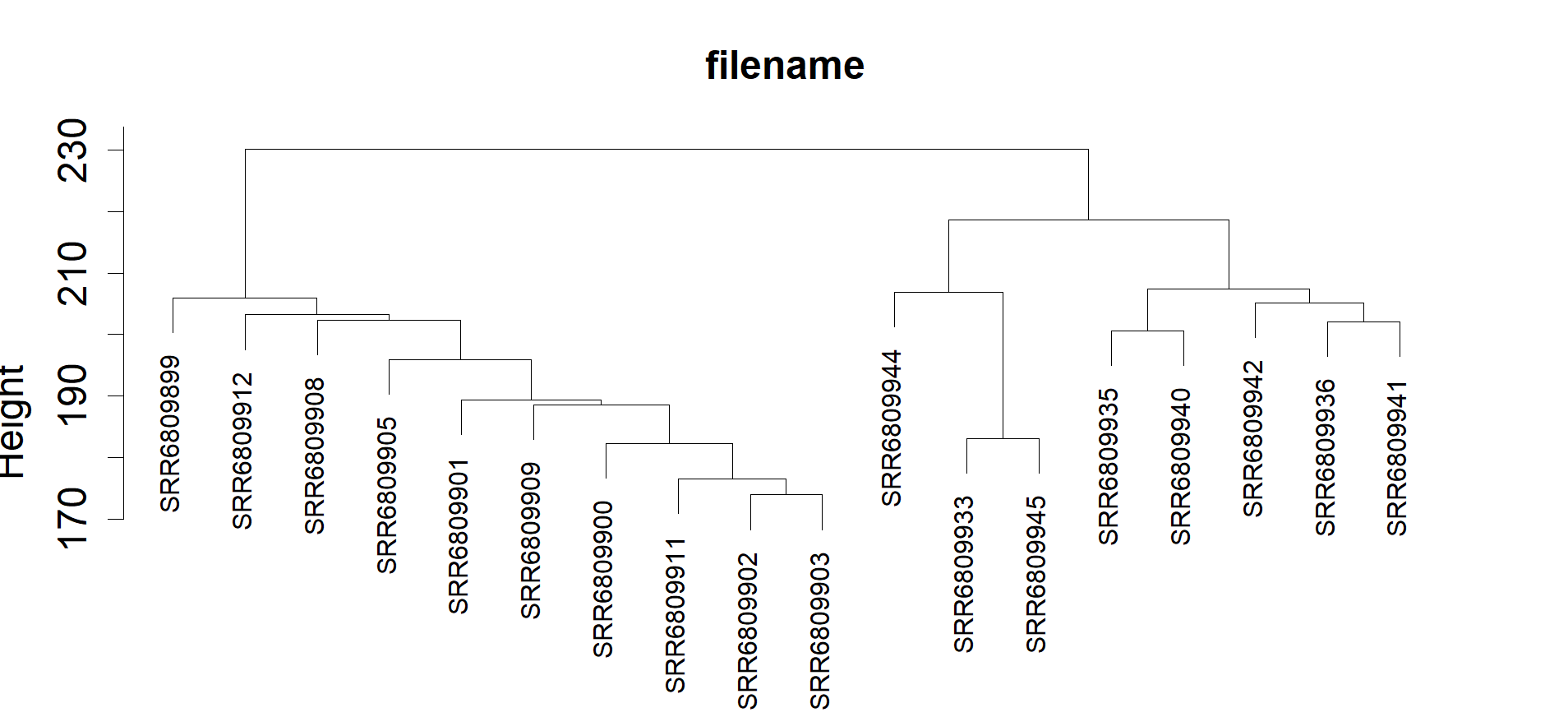
dev.off() # 关闭画图装置上述可以画的是聚类树,效果图如下:
下一期我们做样本间的各种相关图,期待与您下次再见~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~



